© 2011 г. Г. А. Денисова1, Б. А. Малярчук1, М. В. Деренко1, О. А. Кравцова2
1 Учреждение Российской академии наук Институт биологических проблем Севера Дальневосточного отделения РАН, Магадан 685000
Нуклеотидные последовательности ГВС1 мтДНК определены с помощью метода капиллярного секвенирования ДНК с использованием генетического анализатора ABI 3130 ("Applied Bio-systems", Foster City, США). Анализ результатов секвенирования производился с использованием пакета программ SeqScape v. 2.5 ("Applied Biosys-tems", Foster City, США). Полиморфизм однонук-леотидных замен, определяющих гаплогруппы мтДНК, исследовали с помощью рестрикцион-ного анализа [8, 9]. Для расчета параметров генетического разнообразия и проведения анализа межпопуляцион-ной дифференциации (AMOVA) использовали программы пакета ARLEQUIN 3.11 [10]. Корреляции между матрицами генетических, географических и лингвистических дистанций оценивали с использованием теста Мантеля [11] (1000 пермутаций), также реализованного в пакете программ ARLEQUIN 3.11. В качестве генетических дистанций использовали матрицу попарных значений индексов генетической дифференциации (FST) между популяциями. Матрица лингвистических расстояний между популяциями была построена согласно подходу, предложенному в работе ВА. Степанова [12]: расстояние между популяциями, принадлежащими к разным языковым семьям, принято за 1.00, в случае принадлежности пары популяций к одной лингвистической семье — 0.50, одной группе внутри языковой семьи — 0.25. Расстояния в км между населенными пунктами определялись с помощью программы Calculator for Distances between Geographical Locations (http://www.go.ednet.ns.ca/~larry/bsc/jslatlng. html). Матрицу попарных значений индексов генетической дифференциации (FST) использовали для реконструкции филогенетических взаимоотношений исследованных популяций поволжских татар друг с другом, а также с населением Волго-Уральского региона с помощью факторного анализа (метод главных компонент), реализованного в пакете программ STATISTICA 6.0.
Для сравнительного анализа использовали литературные данные о полиморфизме ГВС1 мтДНК населения Волго-Уральского региона: татар, башкир, чувашей, марийцев, мордвы, коми-пермяков и коми-зырян, удмуртов [5, 6], эстонцев и карелов [13], а также трех выборок русского населения Белгородской, Нижегородской и Новгородской областей [14, 15] (табл. 1).
На основании данных об изменчивости ГВС1 и полиморфизме рестрикционных сайтов, определяющих гаплогруппы мтДНК, нами охарактеризованы митохондриальные генофонды двух популяций поволжских татар: из Буинского и Азнакаевского районов Татарстана (табл. 2).
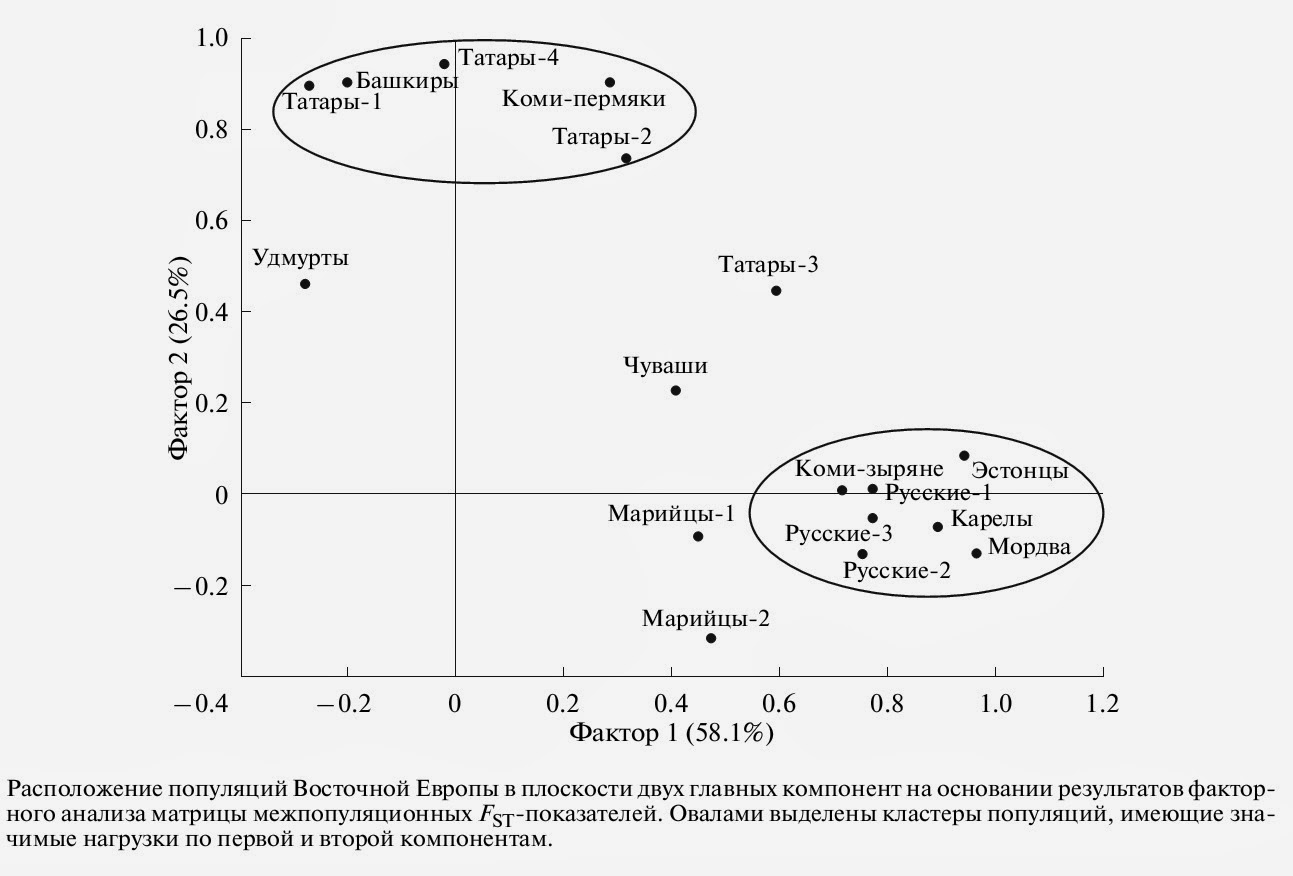
Основываясь на анализе попарных нуклеотид-ных различий между гаплотипами ГВС1 мтДНК, значение межэтнической дифференциации населения Восточной Европы составило 1.8% (P < 0.001). Это значение намного выше, чем для этнических групп Центральной Европы (FST = 0.1%, P = 0.1 для словаков, чехов, поляков, австрийцев, южных немцев, боснийцев и словенцев [16]), но существенно ниже, чем у населения Центральной Азии (FST = 2.34%, P < 0.0001)
[17] и Южной Сибири (FST = 2.86%, P < 0.001) [18]. Между тем степень межпопуляционной дифференциации поволжских татар (FST = 0.33%, P = 0.04) вполне соответствует таковой у русского населения Восточной Европы (FST = 0.35%, P = 0.009) [19]. Для выявления факторов, определяющих характер генетических взаимоотношений между популяциями Восточной Европы, нами проведен факторный анализ, выполненный с помощью метода главных компонент (рисунок). Три первые главные компоненты отражают 92.3% всей изменчивости. На долю первой компоненты приходится 58%, второй — 26.5%, третьей — 7.8% суммарной изменчивости. В пространстве первых двух компонент можно выделить два крупных кластера популяций. Первый объединяет популяции коми-зырян, мордвы, карелов, эстонцев и все три выборки русских. Все популяции этого кластера имеют значимые нагрузки по первой компоненте (значимыми считали нагрузки, абсолютное значение которых превышало 0.7). Второй кластер образуют три выборки татар (татары-1, -2 и -4), башкиры и коми-пермяки, имеющие значимые нагрузки для второго фактора. По третьей компоненте объединяются друг с другом обе выборки марийцев и чуваши.
Для определения факторов, которые в наибольшей степени определяют структуру генетического разнообразия, популяции Восточной Европы были сгруппированы согласно этнолингвистической классификации, а также в соответствии с географическим расположением (табл. 5).
1 Учреждение Российской академии наук Институт биологических проблем Севера Дальневосточного отделения РАН, Магадан 685000
2 Казанский государственный университет им. В.И. Ульянова-Ленина кафедра биохимии, Казань 420008
Получены данные об изменчивости митохондриальной ДНК (мтДНК) в двух популяциях поволжских татар, представляющих население Буинского и Азнакаевского районов Республики Татарстан. Сравнительный анализ данных об изменчивости мтДНК в популяциях Восточной Европы показал, что поволжские татары характеризуются низким уровнем межпопуляционной дифференциации (FST = 0.33%), в то время как уровень межэтнической дифференциации в Восточной Европе составляет 1.8%. Обнаружено генетическое сходство татар восточных районов Татарстана с башкирами, а татар из западных районов с чувашами, с которыми они граничат территориально. Выявлена положительная связь генетической структуры населения Восточной Европы с лингвистической принадлежностью изученных этнических групп.Предполагается, что поволжские татары произошли от тюркоязычных племен, которые расселились в раннем средневековье на территории Средней Волги и Нижнего Прикамья, включив в свой состав более раннее, видимо, финно-угорское и скифо-сарматское население этих территорий Восточной Европы [1, 2]. Само расположение Поволжья на стыке между Европой и Азией, между степями Юга и лесами Севера, издревле способствовало межэтническим контактам и стало главной причиной большой сложности этнической истории населения этого региона. В антропологическом отношении примерно 80% поволжских татар характеризуются европеоидными признаками и примерно 20% — монголоидными, и в целом они являются представителями субуральского типа [3], хотя, по-видимому, различные территориальные группы поволжских татар могут обладать антропологическим своеобразием вследствие полиморфизма антропологических признаков [4]. Несмотря на то что татары являются вторым по численности народом Российской Федерации, они относятся к числу малоизученных в генетическом отношении групп населения [5—7].
Исследования изменчивости наследуемой по материнской линии митохондриальной ДНК (мтДНК) показали, что среди популяций Волго-Уральского региона поволжские татары характеризуются достаточно высоким уровнем генетического разнообразия и промежуточными частотами (примерно 12%) гаплогрупп мтДНК, характерных для генофондов населения Восточной Евразии (Сибири, Центральной и Восточной Азии) [5]. Между тем следует отметить, что имеющиеся в литературе данные об изменчивости нуклеотидных последовательностей гипервариабельного сегмента 1 (ГВС1) главной некодирующей области мтДНК были получены у населения только двух районов Республики Татарстан — Альметьевском) и Елабужского [5, 6], и поэтому дальнейшие исследования полиморфизма мтДНК в популяциях поволжских татар представляются вполне целесообразными. В настоящей работе нами представлены данные об изменчивости мтДНК (на уровне нуклеотидных последовательностей ГВС1 и отдельных однонуклеотидных замен кодирующей области, определяющих гапло-группы мтДНК) в двух популяциях поволжских татар — из Буинского и Азнакаевского районов Республики Татарстан, и проводится анализ межпопуляционных взаимоотношений с учетом опубликованных ранее данных об изменчивости ГВС1 мтДНК в популяциях Восточной Европы.
Материалом для исследования служили образцы ДНК, выделенной из крови неродственных индивидов, представляющих две этнографические группы татар: казанские татары и татары-мишари. Выборка казанских татар собрана в Азнакаевском р-не Республики Татарстан (N = 71), а татары-мишари представлены дисперсной выборкой из Буинского р-на Республики Татарстан (N = 126). Материал собран в ходе экспедиционных выездов в 2004—2005 гг. Этническую принадлежность выясняли путем индивидуального анкетирования, учитывая данные до третьего поколения.
Получены данные об изменчивости митохондриальной ДНК (мтДНК) в двух популяциях поволжских татар, представляющих население Буинского и Азнакаевского районов Республики Татарстан. Сравнительный анализ данных об изменчивости мтДНК в популяциях Восточной Европы показал, что поволжские татары характеризуются низким уровнем межпопуляционной дифференциации (FST = 0.33%), в то время как уровень межэтнической дифференциации в Восточной Европе составляет 1.8%. Обнаружено генетическое сходство татар восточных районов Татарстана с башкирами, а татар из западных районов с чувашами, с которыми они граничат территориально. Выявлена положительная связь генетической структуры населения Восточной Европы с лингвистической принадлежностью изученных этнических групп.Предполагается, что поволжские татары произошли от тюркоязычных племен, которые расселились в раннем средневековье на территории Средней Волги и Нижнего Прикамья, включив в свой состав более раннее, видимо, финно-угорское и скифо-сарматское население этих территорий Восточной Европы [1, 2]. Само расположение Поволжья на стыке между Европой и Азией, между степями Юга и лесами Севера, издревле способствовало межэтническим контактам и стало главной причиной большой сложности этнической истории населения этого региона. В антропологическом отношении примерно 80% поволжских татар характеризуются европеоидными признаками и примерно 20% — монголоидными, и в целом они являются представителями субуральского типа [3], хотя, по-видимому, различные территориальные группы поволжских татар могут обладать антропологическим своеобразием вследствие полиморфизма антропологических признаков [4]. Несмотря на то что татары являются вторым по численности народом Российской Федерации, они относятся к числу малоизученных в генетическом отношении групп населения [5—7].
Исследования изменчивости наследуемой по материнской линии митохондриальной ДНК (мтДНК) показали, что среди популяций Волго-Уральского региона поволжские татары характеризуются достаточно высоким уровнем генетического разнообразия и промежуточными частотами (примерно 12%) гаплогрупп мтДНК, характерных для генофондов населения Восточной Евразии (Сибири, Центральной и Восточной Азии) [5]. Между тем следует отметить, что имеющиеся в литературе данные об изменчивости нуклеотидных последовательностей гипервариабельного сегмента 1 (ГВС1) главной некодирующей области мтДНК были получены у населения только двух районов Республики Татарстан — Альметьевском) и Елабужского [5, 6], и поэтому дальнейшие исследования полиморфизма мтДНК в популяциях поволжских татар представляются вполне целесообразными. В настоящей работе нами представлены данные об изменчивости мтДНК (на уровне нуклеотидных последовательностей ГВС1 и отдельных однонуклеотидных замен кодирующей области, определяющих гапло-группы мтДНК) в двух популяциях поволжских татар — из Буинского и Азнакаевского районов Республики Татарстан, и проводится анализ межпопуляционных взаимоотношений с учетом опубликованных ранее данных об изменчивости ГВС1 мтДНК в популяциях Восточной Европы.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследования служили образцы ДНК, выделенной из крови неродственных индивидов, представляющих две этнографические группы татар: казанские татары и татары-мишари. Выборка казанских татар собрана в Азнакаевском р-не Республики Татарстан (N = 71), а татары-мишари представлены дисперсной выборкой из Буинского р-на Республики Татарстан (N = 126). Материал собран в ходе экспедиционных выездов в 2004—2005 гг. Этническую принадлежность выясняли путем индивидуального анкетирования, учитывая данные до третьего поколения.
Нуклеотидные последовательности ГВС1 мтДНК определены с помощью метода капиллярного секвенирования ДНК с использованием генетического анализатора ABI 3130 ("Applied Bio-systems", Foster City, США). Анализ результатов секвенирования производился с использованием пакета программ SeqScape v. 2.5 ("Applied Biosys-tems", Foster City, США). Полиморфизм однонук-леотидных замен, определяющих гаплогруппы мтДНК, исследовали с помощью рестрикцион-ного анализа [8, 9]. Для расчета параметров генетического разнообразия и проведения анализа межпопуляцион-ной дифференциации (AMOVA) использовали программы пакета ARLEQUIN 3.11 [10]. Корреляции между матрицами генетических, географических и лингвистических дистанций оценивали с использованием теста Мантеля [11] (1000 пермутаций), также реализованного в пакете программ ARLEQUIN 3.11. В качестве генетических дистанций использовали матрицу попарных значений индексов генетической дифференциации (FST) между популяциями. Матрица лингвистических расстояний между популяциями была построена согласно подходу, предложенному в работе ВА. Степанова [12]: расстояние между популяциями, принадлежащими к разным языковым семьям, принято за 1.00, в случае принадлежности пары популяций к одной лингвистической семье — 0.50, одной группе внутри языковой семьи — 0.25. Расстояния в км между населенными пунктами определялись с помощью программы Calculator for Distances between Geographical Locations (http://www.go.ednet.ns.ca/~larry/bsc/jslatlng. html). Матрицу попарных значений индексов генетической дифференциации (FST) использовали для реконструкции филогенетических взаимоотношений исследованных популяций поволжских татар друг с другом, а также с населением Волго-Уральского региона с помощью факторного анализа (метод главных компонент), реализованного в пакете программ STATISTICA 6.0.
Для сравнительного анализа использовали литературные данные о полиморфизме ГВС1 мтДНК населения Волго-Уральского региона: татар, башкир, чувашей, марийцев, мордвы, коми-пермяков и коми-зырян, удмуртов [5, 6], эстонцев и карелов [13], а также трех выборок русского населения Белгородской, Нижегородской и Новгородской областей [14, 15] (табл. 1).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
На основании данных об изменчивости ГВС1 и полиморфизме рестрикционных сайтов, определяющих гаплогруппы мтДНК, нами охарактеризованы митохондриальные генофонды двух популяций поволжских татар: из Буинского и Азнакаевского районов Татарстана (табл. 2).
Всего зарегистрировано 27 гаплогрупп мтДНК, из которых более частыми у татар являются гаплогруппы, распространенные в популяциях Западной Евразии (Европы и Западной Азии). Частота восточноевразийского компонента составила 24% в популяции Азнакаевского р-на и 12% в популяции Буинского р-на (табл. 2). По данным Бермишевой и др. [5], частота восточноевразийских вариантов мтДНК у татар Альметьевского и Елабужского районов составляет 12.8%. Таким образом, поволжские татары занимают промежуточное положение среди этнических групп Волго-Уральского региона, характеризующихся высокими (более 20% у башкир и удмуртов) и умеренными частотами (менее 10% у чувашей, марийцев, мордвы) восточноевразийских вариантов мтДНК [5].
Анализ генетического разнообразия нуклеотидных последовательностей ГВС1 мтДНК в популяциях Восточной Европы свидетельствует о том, что показатели генетического разнообразия у татар из Азнакаевского и Буинского районов в целом соответствуют таковым для других популяций Волго-Уральского региона (табл. 3).
В сравнении с двумя другими ранее исследованными выборками поволжских татар [5, 6] следует отметить, что выборка татар из наиболее западного Буинского р-на характеризуется наиболее низкими значениями показателей генетического разнообразия, что может быть отчасти обусловлено более низкой частотой восточноевразийских линий мтДНК, способствующих увеличению внутрипо-пуляционной гетерогенности.
Для анализа популяционной структуры у татар нами исследована межпопуляционная дифференциация на основании данных о распределении как гаплогрупп мтДНК, так и отдельных гапло-типов ГВС1. Анализ показал, что степень межэтнической дифференциации (FST) по гаплогруппам мтДНК составляет в Восточной Европе 2.6% (P = = 0), а межпопуляционная дифференциация у поволжских татар на порядок ниже — 0.26%. При этом все попарные FST- различия между выборками татар были недостоверными. В случае анализа дифференциации на уровне гаплотипов ГВС1 мтДНК значение межпопуляционных различий для четырех выборок поволжских татар составляет 0.33% (P = 0.04) и в ряде случаев отмечаются достоверные FST-различия между отдельными выборками татар при попарных сравнениях (табл. 4). Анализ показал также, что татары из Азнакаевского р-на достоверно не отличаются от башкир, а татары из Буинского р-на от чувашей, что может объясняться географической близостью Азнакаевского р-на (восточные районы Татарстана) к Башкортостану, а Буинского р-на (запад Татарстана) к Чувашии (табл. 4).
[17] и Южной Сибири (FST = 2.86%, P < 0.001) [18]. Между тем степень межпопуляционной дифференциации поволжских татар (FST = 0.33%, P = 0.04) вполне соответствует таковой у русского населения Восточной Европы (FST = 0.35%, P = 0.009) [19]. Для выявления факторов, определяющих характер генетических взаимоотношений между популяциями Восточной Европы, нами проведен факторный анализ, выполненный с помощью метода главных компонент (рисунок). Три первые главные компоненты отражают 92.3% всей изменчивости. На долю первой компоненты приходится 58%, второй — 26.5%, третьей — 7.8% суммарной изменчивости. В пространстве первых двух компонент можно выделить два крупных кластера популяций. Первый объединяет популяции коми-зырян, мордвы, карелов, эстонцев и все три выборки русских. Все популяции этого кластера имеют значимые нагрузки по первой компоненте (значимыми считали нагрузки, абсолютное значение которых превышало 0.7). Второй кластер образуют три выборки татар (татары-1, -2 и -4), башкиры и коми-пермяки, имеющие значимые нагрузки для второго фактора. По третьей компоненте объединяются друг с другом обе выборки марийцев и чуваши.
Для определения факторов, которые в наибольшей степени определяют структуру генетического разнообразия, популяции Восточной Европы были сгруппированы согласно этнолингвистической классификации, а также в соответствии с географическим расположением (табл. 5).
Максимальные значения межгрупповой изменчивости получены при объединении популяций согласно принадлежности клингвистическим подгруппам (0.71%), однако ни в одном из вариантов группировки доля межгрупповых различий не превышала значения межпопуляционных различий внутри групп. Это свидетельствует, во-первых, об отсутствии связи между структурой генетического разнообразия изученного населения Восточной Европы и географическим распределением и, во-вторых, о влиянии, хотя и незначительном, лингвистического фактора на межпопуляционную генетическую дифференциацию.
Результаты исследования корреляций матриц генетических, географических и лингвистических дистанций с помощью теста Мантеля выявили в Восточной Европе наличие достоверной корреляции между матрицей генетических дистанций и лингвистической принадлежностью исследованных популяций (r = 0.147, P = 0.025) (табл. 6).
Коэффициент частной корреляции матриц генетических и лингвистических дистанций при постоянстве географических расстояний также является высоким и достоверным (r = 0.155, P = 0.03), что подтверждает связь структуры генофондов изученных популяций с лингвистической принадлежностью. Между тем положительной достоверной корреляции между генетическими и географическими расстояниями для исследованных популяций Восточной Европы не выявлено и в этом случае. Таким образом, проведенное исследование разнообразия мтДНК позволило получить первые представления об уровне и характере межпопуляционной дифференциации у поволжских татар, что является важным этапом исследований в области молекулярной филогеографии населения Восточной Европы.
1. Халиков А.Х. Происхождение татар Поволжья и Приуралья. Казань: Татарское кн. изд-во, 1978. 160 с.
2. Кузеев Р.Г. Народы Среднего Поволжья и Южного Урала: этногенетический взгляд на историю. М.: Наука, 1992. 345 с.
3. Алексеев В.П. Происхождение народов Восточной Европы (краниологическое исследование). М.: Наука, 1969. 324 с.
4. Газимзянов И.Р. Антропологический облик татар // Татары / Под ред. Р.К. Уразманова, С.В. Чешко. М.: Наука, 2001. С. 35—40.
5. Бермишева М.А., Тамбетс К., Виллемс Р., Хуснутди-нова Э.К. Разнообразие гаплогрупп митохондри-альной ДНК в этнических группах Волго-Ураль-ского региона // Молекуляр. биология. 2002. Т. 36. С. 990—1001.
6. Орехов В.А. Характеристика митотипов представителей трех этнических групп европейской части России: Автореф. дис. ... канд. биол. наук. М.: ИОГен РАН, 2002. 22 с.
7. Кравцова О.А. Молекулярно-генетический анализ древних и современных образцов ДНК: Автореф. дис. . канд. биол. наук. Казань: КГУ, 2006. 24 с.
8. Malyarchuk B.A., Grzybowski T., Derenko M.V. et al. Mitochondrial DNA variability in Poles and Russians // Ann. Hum. Genet. 2002. V. 66. P. 261—283.
9. Derenko M, Malyarchuk B., Grzybowski T. et al. Phylo-geographic analysis of mitochondrial DNA in North
Asian populations // Am. J. Hum. Genet. 2007. V. 81. P. 1025—1041.
10. ExcoffierL., LavalG., SchneiderS. Arlequin ver. 3.0: An integrated software package for population genetics data analysis // Evol. Bioinf. Online. 2005. V. 1. P. 47—50.
11. Mantel N. The detection of disease clustering and a generalized regression approach // Cancer Res. 1967. V. 27. P. 209—220.
12. Степанов В.А. Этногеномика населения Северной Евразии. Томск: Печатная мануфактура, 2002. 244 с.
13. Sajantila A., Lahermo P., Anttinen T. et al. Genes and languages in Europe — an analysis of mitochondrial lineages // Genome Res. 1995. V. 5. P. 42—52.
14. Малярчук Б.А. Изменчивость митохондриального генома человека в аспекте генетической истории славян: Дис. . д-ра биол. наук. Магадан: ИБПС ДВО РАН, 2002. 480 с.
15. Grzybowski T., Malyarchuk B.A., Derenko M.V. et al. Complex interactions of the Eastern and Western Slavic populations with other European groups as revealed by mitochondrial DNA analysis // Forensic Sci. Int. Genet. 2007. V. 1. P. 141—147.
16. Malyarchuk B.A., Perkova M.A., Derenko M.V. et al. Mitochondrial DNA variability in Slovaks, with application to the Roma origin // Ann. Hum. Genet. 2008. V. 72. P. 228—240.
17. Comas D., Plaza S., Wells R.S. et al. Admixture, migrations, and dispersals in Central Asia: evidence from maternal DNA lineages // Eur. J. Hum. Genet. 2004. V. 12. P. 495—504.
18. Derenko M.V., Grzybowski T., Malyarchuk B.A. et al. Diversity of mitochondrial DNA lineages in South Siberia // Ann. Hum. Genet. 2003. V. 67. P. 391—411.
19. Малярчук Б.А. Вклад балтийских славян в популяционно-генетическую дифференциацию русского населения Восточной Европы // Вестник СВНЦ ДВО РАН. 2008. № 3. C. 55—59.
Результаты исследования корреляций матриц генетических, географических и лингвистических дистанций с помощью теста Мантеля выявили в Восточной Европе наличие достоверной корреляции между матрицей генетических дистанций и лингвистической принадлежностью исследованных популяций (r = 0.147, P = 0.025) (табл. 6).
СПИСОК ЛИТЕРАТУРЫ
1. Халиков А.Х. Происхождение татар Поволжья и Приуралья. Казань: Татарское кн. изд-во, 1978. 160 с.
2. Кузеев Р.Г. Народы Среднего Поволжья и Южного Урала: этногенетический взгляд на историю. М.: Наука, 1992. 345 с.
3. Алексеев В.П. Происхождение народов Восточной Европы (краниологическое исследование). М.: Наука, 1969. 324 с.
4. Газимзянов И.Р. Антропологический облик татар // Татары / Под ред. Р.К. Уразманова, С.В. Чешко. М.: Наука, 2001. С. 35—40.
5. Бермишева М.А., Тамбетс К., Виллемс Р., Хуснутди-нова Э.К. Разнообразие гаплогрупп митохондри-альной ДНК в этнических группах Волго-Ураль-ского региона // Молекуляр. биология. 2002. Т. 36. С. 990—1001.
6. Орехов В.А. Характеристика митотипов представителей трех этнических групп европейской части России: Автореф. дис. ... канд. биол. наук. М.: ИОГен РАН, 2002. 22 с.
7. Кравцова О.А. Молекулярно-генетический анализ древних и современных образцов ДНК: Автореф. дис. . канд. биол. наук. Казань: КГУ, 2006. 24 с.
8. Malyarchuk B.A., Grzybowski T., Derenko M.V. et al. Mitochondrial DNA variability in Poles and Russians // Ann. Hum. Genet. 2002. V. 66. P. 261—283.
9. Derenko M, Malyarchuk B., Grzybowski T. et al. Phylo-geographic analysis of mitochondrial DNA in North
Asian populations // Am. J. Hum. Genet. 2007. V. 81. P. 1025—1041.
10. ExcoffierL., LavalG., SchneiderS. Arlequin ver. 3.0: An integrated software package for population genetics data analysis // Evol. Bioinf. Online. 2005. V. 1. P. 47—50.
11. Mantel N. The detection of disease clustering and a generalized regression approach // Cancer Res. 1967. V. 27. P. 209—220.
12. Степанов В.А. Этногеномика населения Северной Евразии. Томск: Печатная мануфактура, 2002. 244 с.
13. Sajantila A., Lahermo P., Anttinen T. et al. Genes and languages in Europe — an analysis of mitochondrial lineages // Genome Res. 1995. V. 5. P. 42—52.
14. Малярчук Б.А. Изменчивость митохондриального генома человека в аспекте генетической истории славян: Дис. . д-ра биол. наук. Магадан: ИБПС ДВО РАН, 2002. 480 с.
15. Grzybowski T., Malyarchuk B.A., Derenko M.V. et al. Complex interactions of the Eastern and Western Slavic populations with other European groups as revealed by mitochondrial DNA analysis // Forensic Sci. Int. Genet. 2007. V. 1. P. 141—147.
16. Malyarchuk B.A., Perkova M.A., Derenko M.V. et al. Mitochondrial DNA variability in Slovaks, with application to the Roma origin // Ann. Hum. Genet. 2008. V. 72. P. 228—240.
17. Comas D., Plaza S., Wells R.S. et al. Admixture, migrations, and dispersals in Central Asia: evidence from maternal DNA lineages // Eur. J. Hum. Genet. 2004. V. 12. P. 495—504.
18. Derenko M.V., Grzybowski T., Malyarchuk B.A. et al. Diversity of mitochondrial DNA lineages in South Siberia // Ann. Hum. Genet. 2003. V. 67. P. 391—411.
19. Малярчук Б.А. Вклад балтийских славян в популяционно-генетическую дифференциацию русского населения Восточной Европы // Вестник СВНЦ ДВО РАН. 2008. № 3. C. 55—59.

+%D0%B3%D0%B0%D0%BF%D0%BB%D0%BE%D0%B3%D1%80%D1%83%D0%BF%D0%BF+%D0%BC%D1%82%D0%94%D0%9D%D0%9A+%D1%83+%D0%BF%D0%BE%C2%AC%D0%B2%D0%BE%D0%BB%D0%B6%D1%81%D0%BA%D0%B8%D1%85+%D1%82%D0%B0%D1%82%D0%B0%D1%80.jpg)

+%D0%BF%D0%BE+%D0%B4%D0%B0%D0%BD%D0%BD%D1%8B%D0%BC+%D0%BE%D0%B1+%D0%B8%D0%B7%D0%BC%D0%B5%D0%BD%D1%87%D0%B8%D0%B2%D0%BE%D1%81%D1%82%D0%B8+%D0%93%D0%92%D0%A11+%D0%BC%D1%82%D0%94%D0%9D.jpg)

